Het arrangement Genomics is gemaakt met Wikiwijs van Kennisnet. Wikiwijs is hét onderwijsplatform waar je leermiddelen zoekt, maakt en deelt.
- Auteur
- Laatst gewijzigd
- 09-06-2021 09:28:26
- Licentie
-
Dit lesmateriaal is gepubliceerd onder de Creative Commons Naamsvermelding-GelijkDelen 4.0 Internationale licentie. Dit houdt in dat je onder de voorwaarde van naamsvermelding en publicatie onder dezelfde licentie vrij bent om:
- het werk te delen - te kopiëren, te verspreiden en door te geven via elk medium of bestandsformaat
- het werk te bewerken - te remixen, te veranderen en afgeleide werken te maken
- voor alle doeleinden, inclusief commerciële doeleinden.
Meer informatie over de CC Naamsvermelding-GelijkDelen 4.0 Internationale licentie.
Aanvullende informatie over dit lesmateriaal
Van dit lesmateriaal is de volgende aanvullende informatie beschikbaar:
- Toelichting
- De Kennisbanken bevatten de theorie bij de opdrachten.
- Leerinhoud en doelen
- Biologie;
- Eindgebruiker
- leerling/student
- Moeilijkheidsgraad
- gemiddeld



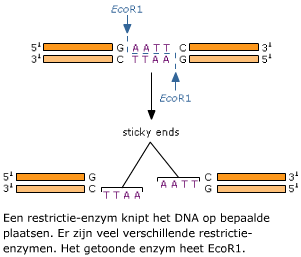
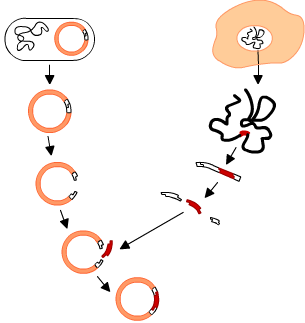 Elke bacterie heeft behalve een groot chromosoom ook nog kleine cirkelvormige DNA-moleculen, de plasmiden. Die plasmiden kunnen ze vrij gemakkelijk van de ene naar de andere bacterie overbrengen.
Elke bacterie heeft behalve een groot chromosoom ook nog kleine cirkelvormige DNA-moleculen, de plasmiden. Die plasmiden kunnen ze vrij gemakkelijk van de ene naar de andere bacterie overbrengen.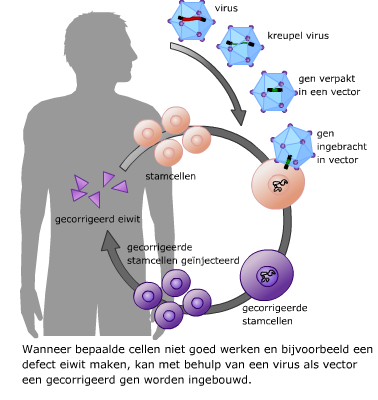 Virussen bouwen hun erfelijk materiaal in dat van de gastheer in.
Virussen bouwen hun erfelijk materiaal in dat van de gastheer in.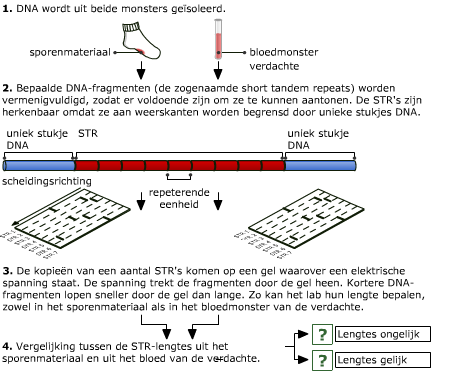
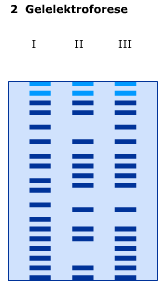
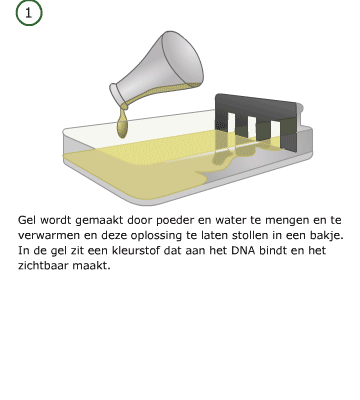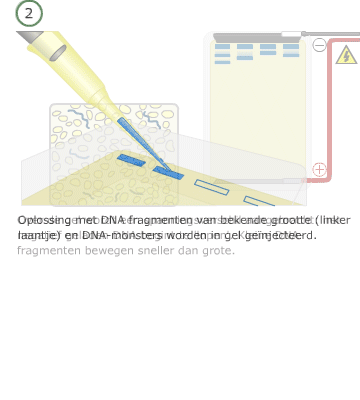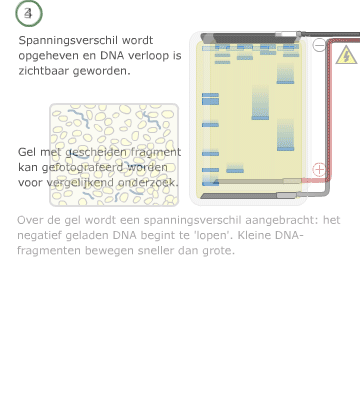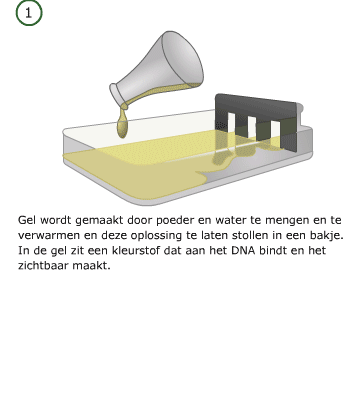 Bij gelelektroforese worden de stukken DNA die bij PCR zijn gemaakt, zichtbaar gemaakt. Eerst wordt een gel gemaakt, waarin vervolgens het mengsel van DNA dat bij PCR gemaakt is wordt geïnjecteerd. Vervolgens wordt er elektrische spanning over de gel gezet. De stukken DNA bewegen dan van – naar +. De lange stukken zijn langzamer dan de korte stukken. Zo worden de stukken DNA van elkaar gescheiden.
Bij gelelektroforese worden de stukken DNA die bij PCR zijn gemaakt, zichtbaar gemaakt. Eerst wordt een gel gemaakt, waarin vervolgens het mengsel van DNA dat bij PCR gemaakt is wordt geïnjecteerd. Vervolgens wordt er elektrische spanning over de gel gezet. De stukken DNA bewegen dan van – naar +. De lange stukken zijn langzamer dan de korte stukken. Zo worden de stukken DNA van elkaar gescheiden.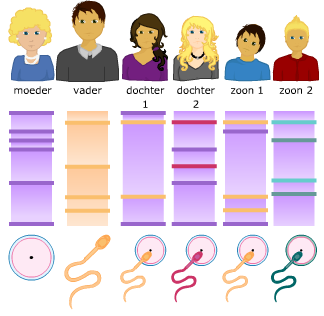 Als op het DNA van iemand een PCR wordt toegepast met bepaalde primers, ontstaan er stukken DNA van een bepaalde lengte. Deze stukken kunnen worden gescheiden door middel van elektroforese. Er ontstaat nu een analyse product dat voor ieder uniek is. Als dit unieke analyseproduct zichtbaar wordt gemaakt, krijg je een patroon van banden. Dit patroon noemt men wel een fingerprint, omdat het net zo uniek is als een vingerafdruk.
Als op het DNA van iemand een PCR wordt toegepast met bepaalde primers, ontstaan er stukken DNA van een bepaalde lengte. Deze stukken kunnen worden gescheiden door middel van elektroforese. Er ontstaat nu een analyse product dat voor ieder uniek is. Als dit unieke analyseproduct zichtbaar wordt gemaakt, krijg je een patroon van banden. Dit patroon noemt men wel een fingerprint, omdat het net zo uniek is als een vingerafdruk.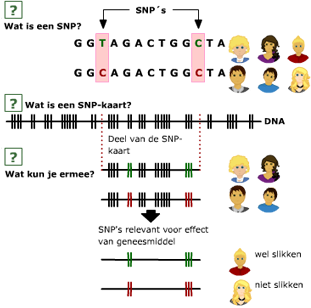 99,9% van het DNA is bij alle mensen hetzelfde. Dat is ook wel begrijpelijk, natuurlijke selectie maakt dat in genen die coderen voor eiwitten niet veel variatie mogelijk is. Niet-coderende stukken DNA variëren echter van persoon tot persoon. Men noemt deze stukjes DNA polymorfismen (= veel vormen). Ongeveer 40 procent van het niet-coderende DNA bestaat uit zogenaamd repetitief DNA. Dat zijn korte DNA-sequenties van maximaal 60 basenparen lang die ettelijke keren herhaald worden. Zeer korte repetitieve sequenties
99,9% van het DNA is bij alle mensen hetzelfde. Dat is ook wel begrijpelijk, natuurlijke selectie maakt dat in genen die coderen voor eiwitten niet veel variatie mogelijk is. Niet-coderende stukken DNA variëren echter van persoon tot persoon. Men noemt deze stukjes DNA polymorfismen (= veel vormen). Ongeveer 40 procent van het niet-coderende DNA bestaat uit zogenaamd repetitief DNA. Dat zijn korte DNA-sequenties van maximaal 60 basenparen lang die ettelijke keren herhaald worden. Zeer korte repetitieve sequenties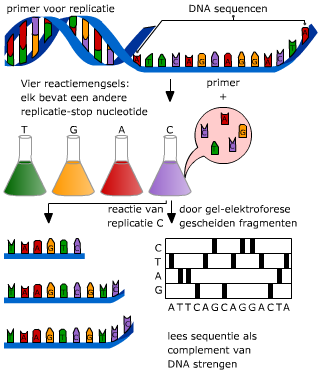 Om de exacte volgorde van de nucelotiden in een stuk DNA vast te stellen, maakt men gebruik van een techniek die sequencen heet.
Om de exacte volgorde van de nucelotiden in een stuk DNA vast te stellen, maakt men gebruik van een techniek die sequencen heet.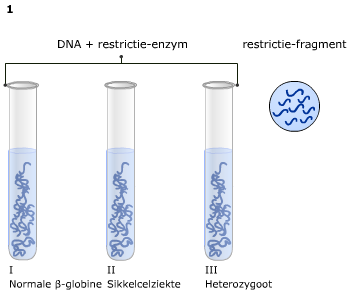
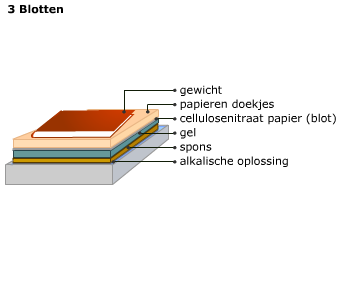
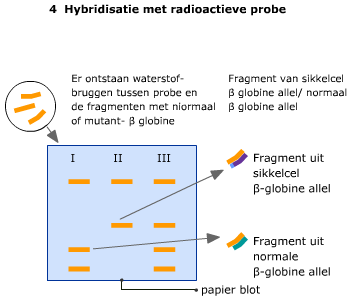
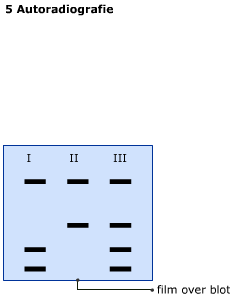
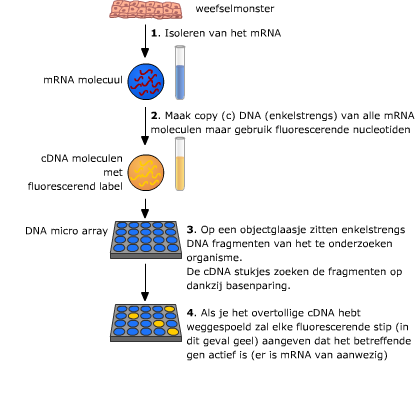 DNA micro-array (DNA chip) wordt gebruikt om te bepalen welke genen worden afgelezen onder bepaalde condities.
DNA micro-array (DNA chip) wordt gebruikt om te bepalen welke genen worden afgelezen onder bepaalde condities.